Kita akan menjelaskan prosedur untuk mengamati proses pertumbuhan kristal es dan pelelehan secara langsung, serta memperkirakan titik leleh es menggunakan simulasi komputer.
0. Pengaturan Lingkungan
Kita akan melakukan perhitungan di cloud.
0-0 Tentang dokumen ini
Bagian yang ditampilkan seperti di bawah ini, kecuali dinyatakan lain, menunjukkan perintah yang diketik di Terminal.
cd MeltingPoint0-1 Persiapan untuk menggunakan cloud
- Kami akan membagikan akun untuk login ke server komputasi (Universitas Okayama atau Amazon EC2). Jika menggunakan komputer Universitas Okayama, siapkan koneksi VPN terlebih dahulu dan pastikan Anda dapat terhubung. (→ 9. Lampiran)
- Dari ikon di sebelah kiri jendela VSCode
 , tambahkan ekstensi berikut:
, tambahkan ekstensi berikut:
- Remote SSH
- Hubungkan ke alamat IP yang ditentukan. Tekan tombol di kiri bawah jendela VSCode
 . (Akan ada beberapa pertanyaan.)
. (Akan ada beberapa pertanyaan.) - Siapkan sumber program di komputer cloud. Tekan
Clone Git Repository...di tengah jendela VSCode, ketika munculClone from GitHubtekan enter, lalu tentukanvitroid/gromacs-usecases. Ketika ditanya lokasi penyimpanan, tekan enter. Langkah ini tidak diperlukan untuk login kedua dan seterusnya. - Buka folder
MeltingPointdi dalam folder tempat sumber disimpan.
0-2 Persiapan komputer lokal
- Kita akan menggunakan alat untuk memplot data. Gunakan Igor Pro, gnuplot, atau ngraph jika Anda bisa menggunakannya. Jika tidak ada, Anda bisa menggunakan Excel, Numbers, atau Google Spreadsheet sebagai alternatif. Namun, kami tidak akan menjelaskan cara menggunakan masing-masing program di sini.
- Instal terlebih dahulu alat visualisasi hasil simulasi VMD.
1. Memilih model molekul air
Model molekul adalah aproksimasi bentuk dan interaksi molekul menggunakan fungsi sederhana. Sebenarnya, gaya yang bekerja antar molekul dan cara ikatan melibatkan keadaan elektron, dan hanya dapat diperoleh dengan menghitung orbit elektron (yang terdistribusi seperti awan di sekitar inti atom) secara akurat, tetapi dalam simulasi molekuler, untuk mengurangi jumlah perhitungan, panjang ikatan ditetapkan atau distribusi elektron diperkirakan dengan muatan titik.
Ada lebih dari 100 jenis model molekul air yang digunakan dalam simulasi komputer. Ini karena konsep aproksimasi yang berbeda. Sebagian besar dirancang untuk mereproduksi sifat-sifat air cair dan larutan pada suhu kamar, sehingga tidak cocok untuk mengamati transisi fasa kristalisasi. (Titik lelehnya jauh menyimpang dari 273 K)
Di sini, kita akan menggunakan model TIP4P/Ice yang baru-baru ini dikembangkan dan memiliki keandalan tinggi. Titik leleh model ini pada tekanan 1 atmosfer dikatakan 270 K [^1], dan sering digunakan untuk mempelajari perilaku air, es, dan hidrat (kristal terhidrasi) pada suhu di bawah suhu kamar.
Bukankah titik leleh es sudah diketahui dari eksperimen? Benar sekali. Tapi yang ingin kita ketahui adalah titik leleh model molekul air yang digunakan dalam simulasi komputer. Jika kualitas model buruk, titik lelehnya tidak akan sesuai dengan nilai eksperimen. Tujuan simulasi molekuler bukan untuk mendapatkan data yang sesuai dengan eksperimen. Nilainya terletak pada mendapatkan informasi yang tidak terlihat dalam eksperimen sambil menjamin reproduksi eksperimen. Jika modelnya membatasi sifat-sifat seperti titik leleh dengan baik, dapat dianggap bahwa gerakan molekulpun direproduksi dengan cukup akurat. Jika demikian, kita dapat mempercayai hasil pengamatan langsung tentang bagaimana air membeku dari antarmuka padat-cair (yang tidak terlihat dalam eksperimen).
Untuk detail bentuk TIP4P/Ice, lihat tautan ini.
Meskipun molekul air terdiri dari 3 atom, ciri khas model air empat titik adalah penambahan satu titik interaksi tambahan untuk mendekati interaksi antara titik-titik massa secara aproksimasi.
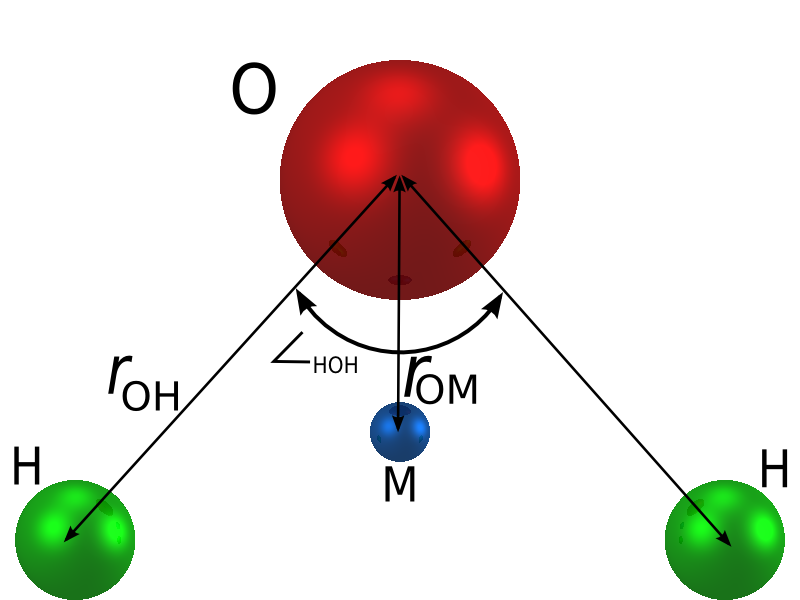
Model molekul TIP4P/Ice tertulis dalam ice.top.
; taken from http://www.sklogwiki.org/SklogWiki/index.php/GROMACS_topology_file_for_the_TIP4P/Ice_model
[ defaults ]
; nbfunc comb-rule gen-pairs fudgeLJ fudgeQQ
1 2 no 1.0 1.0
[atomtypes]
;name mass charge ptype sigma epsilon
IW 0 0.000 D 0.0 0.0
OWT4 15.99940 0.000 A 0.31668 0.88211
HW 1.00800 0.000 A 0.00000E+00 0.00000E+00
...File data ini berisi sekitar 50 baris, dan berisi informasi berikut:
- Berapa banyak titik yang digunakan untuk mendekati satu molekul air.
- Jarak relatif titik-titik dalam molekul.
- Bagaimana titik-titik berinteraksi satu sama lain (gaya Coulomb dan Lennard-Jones).
- Apakah molekul diperlakukan sebagai benda fleksibel dengan titik-titik yang dihubungkan oleh pegas, atau sebagai benda kaku yang tidak berubah bentuk.
Untuk TIP4P/Ice:
- Ada 4 titik interaksi.
- Jarak H-H sekitar 0,15 nm, jarak O-H sekitar 0,1 nm.
- 3 titik berinteraksi Coulomb, 1 titik lainnya berinteraksi Lennard-Jones.
- Diperlakukan sebagai benda kaku.
2. Menempatkan molekul
Bagaimana kita menempatkan molekul di awal sangat mempengaruhi keberhasilan simulasi selanjutnya.
Ketika memperkirakan titik leleh, jika kita memilih konfigurasi awal di mana semua molekul tersusun dalam struktur kristal es, es tidak akan meleleh bahkan jika suhu dinaikkan 10 K atau 20 K di atas titik leleh (fenomena pemanasan berlebih). Ini karena waktu yang dibutuhkan untuk terjadinya kerusakan struktur secara kebetulan, yang menjadi pemicu awal pelelehan, terlalu lama. Tentu saja, jika waktu perhitungan mendekati tak terbatas, es akan mulai meleleh pada suhu di atas titik leleh, tetapi perhitungan selama itu tidak mungkin dilakukan dengan komputer saat ini.
Demikian pula, jika kita mulai dari keadaan cairan sempurna, akan membutuhkan waktu yang sangat lama untuk kristal terbentuk secara alami, berapa pun suhu diturunkan. (fenomena pendinginan berlebih)
Oleh karena itu, ketika ingin memperkirakan titik leleh, kita mempersiapkan keadaan di mana es dan air sudah ada bersama. Dengan cara ini, jika suhu yang ditetapkan lebih tinggi dari titik leleh, es akan perlahan-lahan meleleh, dan jika lebih rendah, kita akan melihat pertumbuhan es.
Konfigurasi awal disiapkan dengan langkah-langkah berikut:
- Gunakan alat GenIce untuk membuat struktur kristal es. Kristal dibuat dalam bentuk balok panjang dengan rasio 1:1:2.
- ”Tetapkan” setengah dari molekul air. Ini berarti mereka tidak bisa bergerak dari posisi awal mereka.
- Naikkan suhu hingga 800 K dan lakukan simulasi dinamika molekuler singkat. Bagian yang ditetapkan akan mempertahankan strukturnya, tetapi molekul air lainnya akan segera meleleh.
- Kembalikan suhu dan lepaskan fiksasi. Ini menghasilkan struktur awal dengan setengah cairan dan setengah padat.
- Lakukan simulasi selama beberapa waktu pada sekitar 270 K (titik leleh) dengan fiksasi dilepas untuk merelaksasi struktur.
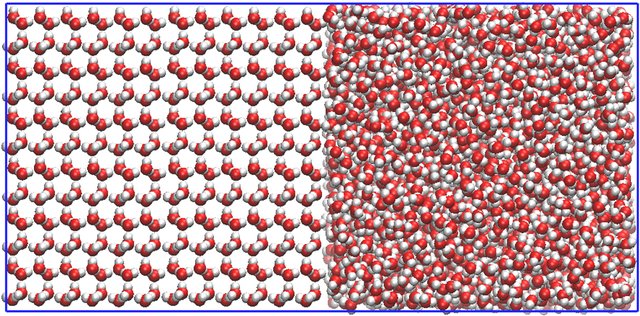
Es yang setengah meleleh. Setengah bagian kiri ditetapkan dan tidak bergerak. [^3] Karena kondisi batas periodik, ujung kiri dan kanan, atas dan bawah, depan dan belakang masing-masing terhubung dan molekul dapat keluar masuk.
Berikut adalah langkah-langkah spesifik:
2-1 Membuat kristal es
GenIce adalah alat untuk menghasilkan berbagai struktur kristal es. Kita akan menggunakannya untuk menghasilkan es Ih (es I heksagonal, jenis es yang paling umum ditemui).
Pertama, buka terminal.
Instal genice2 terlebih dahulu. Ketik yang berikut di terminal:
pip3.11 install numpy genice2Gunakan genice2 yang telah diinstal untuk menghasilkan struktur es.
genice2 1h -r 3 3 6 -w tip4p > ice.groOpsi -r menentukan berapa banyak sel satuan yang akan disusun dalam arah x, y, dan z. Opsi -w menentukan jenis model molekul air.
Di akhir ice.gro, ukuran kotak ditulis dalam satuan nm seperti ini:
...
863ICE HW1 3450 2.178 1.983 5.379
863ICE HW2 3451 2.220 2.107 5.303
863ICE MW 3452 2.217 2.020 5.310
864ICE OW 3453 1.694 1.648 5.310
864ICE HW1 3454 1.740 1.620 5.231
864ICE HW2 3455 1.697 1.744 5.306
864ICE MW 3456 1.700 1.657 5.300
2.34685163 2.20607179 5.42191111Ini adalah sel dengan dimensi 2,3 nm, 2,2 nm, dan 5,4 nm dalam arah sumbu x, y, dan z, sedikit lebih panjang dalam arah z.
Mari kita visualisasikan struktur ini untuk sementara. Klik kanan ice.gro di browser file di sisi kiri jendela VSCode dan unduh. Buka file tersebut dengan VMD.
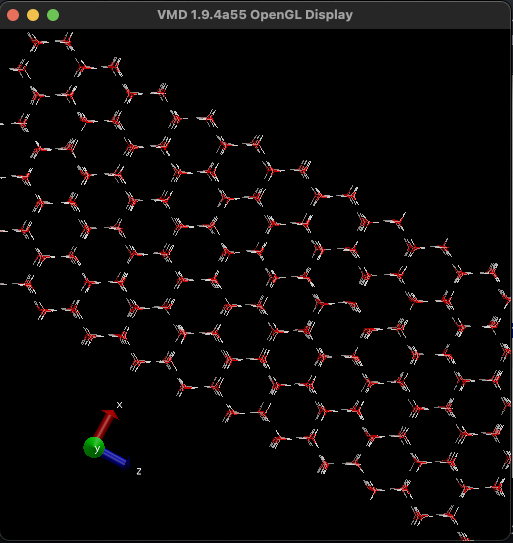
2-2 Menetapkan molekul
Gromacs memiliki fitur untuk membuat atom tidak bergerak dengan menentukan nomor atomnya. Karena akan merepotkan untuk menentukan secara manual, kita akan menggunakan skrip Python untuk menetapkan hanya atom-atom dengan koordinat Z kurang dari 2,7 nm.
2-2-1 Memberi indeks pada atom
Perintah make_ndx dari Gromacs menghasilkan file .ndx yang mengklasifikasikan atom berdasarkan jenisnya.
gmx make_ndx -f ice.gro -o ice.ndxJalankan ini dan tekan q ketika prompt ditampilkan untuk keluar. ice.ndx akan berisi konten seperti berikut:
[ System ]
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15
16 17 18 19 20 21 22 23 24 25 26 27 28 29 30
31 32 33 34 35 36 37 38 39 40 41 42 43 44 45
...File ini mengelompokkan atom-atom. Grup System berisi semua atom.
Mari kita siapkan grup FIX baru yang berisi daftar atom yang ingin kita tetapkan dengan cara yang sama.
2-2-2 Menambahkan indeks atom yang ditetapkan
Tambahkan indeks atom yang ingin ditetapkan ke akhir file ini.
python3 zfix.py 0.0 2.7 < ice.gro >> ice.ndxIni akan menambahkan baris-baris berikut ke akhir file ice.ndx:
...
[FIX]
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21
22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40
41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59
...2-3 Melelehkan setengah sisanya
Kita akan menaikkan suhu hingga 800 K sambil menjaga volume tetap. Biasanya ini akan menyebabkan pendidihan, tetapi karena tidak bisa mengembang, air hanya akan menjadi cair. Langkah ini hanya untuk merusak struktur, jadi tidak perlu dijalankan terlalu lama.
Eksekusi metode dinamika molekuler di Gromacs selalu terdiri dari tiga langkah:
- “Kompilasi” file konfigurasi untuk mengubahnya menjadi format data yang mudah dibaca oleh Gromacs.
- Membaca data tersebut dan menjalankan simulasi dinamika molekuler utama (mdrun).
- Mengubah data yang dihasilkan menjadi format yang mudah dibaca manusia.
2-3-1 Kompilasi
File dengan ekstensi .mdp berisi detail tentang bagaimana melakukan dinamika molekuler. Penjelasan untuk masing-masing parameter ditulis sebagai komentar dalam file fix.mdp. Hampir semua informasi selain konfigurasi molekul dan interaksi, seperti waktu perhitungan, interval perekaman, pengaturan suhu, pengaturan tekanan, dll., ditulis dalam .mdp. Dalam kasus fix.mdp, selain itu juga ada instruksi untuk menetapkan grup atom FIX yang ditentukan dalam file .ndx.
Kompilasi ini dengan perintah berikut untuk membuat file .tpr:
gmx grompp -maxwarn 1 -f fix.mdp -p ice.top -c ice.gro -n ice.ndx -o fixed.tprIni akan membuat file fixed.tpr yang akan dibaca oleh Gromacs.
2-3-2 Eksekusi
Sekarang kita akan menjalankan simulasi dengan membaca 00001.tpr yang dibuat di 2-3-1!
gmx mdrun -deffnm fixedKetika ini dijalankan, banyak file lain yang dimulai dengan fixed. akan dihasilkan. (-deffnm kemungkinan adalah singkatan dari default file name, yang menginstruksikan untuk menambahkan fixed. ke semua file output yang tidak ditentukan secara eksplisit.)
fixed.cpt: File checkpoint, diperlukan untuk melanjutkan perhitungan.fixed.edr: File yang berisi informasi statistik seperti suhu dan tekanan.fixed.trr: Data koordinat dan kecepatan atom (disebut trajektori).fixed.log: Catatan lainnya. Waktu eksekusi juga dicatat dalam file ini.
Di bagian akhir fixed.log, informasi waktu eksekusi dicatat seperti berikut:
starting mdrun 'water TIP4P/Ice'
50000 steps, 100.0 ps.
Writing final coordinates.
Core t (s) Wall t (s) (%)
Time: 792.815 99.102 800.0
(ns/day) (hour/ns)
Performance: 87.185 0.275Ini adalah kasus yang dijalankan di MacBook Air, dan kita dapat melihat bahwa:
- Menggunakan 800% CPU (menggunakan 8 core)
- 50000 langkah dijalankan. Karena 1 langkah adalah 0,002 ps, simulasi 100 ps telah dilakukan.
- Waktu yang dibutuhkan adalah 100 detik. Pada kecepatan ini, perhitungan untuk 90 ns dapat dilakukan dalam satu hari.
2-3-3 Konfirmasi
Mari kita visualisasikan gerakan molekul menggunakan alat visualisasi hasil simulasi VMD.
Pertama, kita akan mengubah konfigurasi molekul yang diperoleh dari perhitungan dinamika molekuler menjadi format yang mudah divisualisasikan.
gmx trjconv -f fixed.trr -s fixed.tpr -pbc whole -o fixed.groKetika diminta input, tekan 0 dan kemudian enter. (Ini akan memvisualisasikan seluruh Sistem)
Ini akan membuat file fixed.gro. Ini adalah file yang cukup besar.
- Di VSCode, klik kanan
fixed.grodan unduh. - Buka VMD.
- Seret dan lepas file yang diunduh ke jendela Utama VMD.
Anda akan melihat bahwa molekul di bagian yang ditetapkan sama sekali tidak bergerak. Di sisi lain, bagian yang tidak ditetapkan menjadi benar-benar acak.
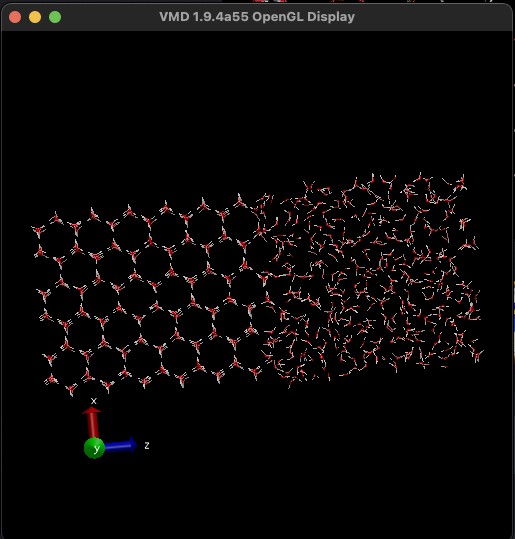
2-3-4 Relaksasi
Kita akan menurunkan suhu, melepaskan fiksasi atom, dan membiarkan struktur berelaksasi. Pada saat ini, kita juga akan memungkinkan volume berubah, dan beralih ke simulasi tekanan konstan.
Seperti sebelumnya, pengaturan simulasi ditulis dalam .mdp. Kali ini kita akan menggunakan relax.mdp.
Anda dapat menggunakan fitur perbandingan file VSCode untuk melihat perbedaan antara relax.mdp dan fix.mdp.
Ada tiga perubahan:
- Suhu diturunkan menjadi 270 K.
- Beralih dari volume konstan ke tekanan konstan.
- Fiksasi molekul dilepaskan.
Mari kita kompilasi lagi menggunakan ini dan jalankan.
gmx grompp -maxwarn 1 -f relax.mdp -p ice.top -c fixed.tpr -t fixed.cpt -o relaxed.tpr
gmx mdrun -deffnm relaxedSeperti di 2-3-3, buatlah relaxed.gro dan amati dengan VMD. Kali ini, Anda akan melihat bahwa molekul di bagian es juga bergetar sedikit.
gmx trjconv -f relaxed.trr -s relaxed.tpr -pbc whole -o relaxed.gro3. Melakukan simulasi pada suhu target
Menggunakan struktur yang dibuat di 2. sebagai konfigurasi awal, kita akan melakukan simulasi jangka panjang pada berbagai suhu.
Target tekanan adalah 1 atmosfer, dan waktu simulasi adalah 3-10 ns. Dalam simulasi ini, kita menggunakan dt=0.002 ps, jadi 1 ns setara dengan 500.000 langkah.
Di sini, sebagai contoh, kita akan menjelaskan perhitungan pada 250 K. Ini 20 K lebih rendah dari titik leleh TIP4P/Ice [^1], jadi es diharapkan akan segera tumbuh. (Untuk suhu selain 250 K, silakan sesuaikan sesuai kebutuhan.)
3-1 Persiapan lingkungan kerja
Buka file pengaturan perhitungan berkelanjutan continue.mdp di VSCode, ubah pengaturan suhu menjadi 250 K seperti yang ditunjukkan di bawah ini, dan simpan dengan nama file yang berbeda sebagai T250.mdp.
...
ref_t = xxxx ; Suhu sistem. Satuan dalam K.
...Juga, di bagian awal T250.mdp, ada instruksi untuk jumlah langkah perhitungan. Jumlah langkah yang diperlukan berbeda untuk suhu rendah dan tinggi. Untuk saat ini, kita ingin melakukan simulasi 3 ns untuk 280 K ke atas, dan 10 ns untuk di bawahnya, tetapi mengingat kemungkinan perhitungan tidak selesai dalam waktu kuliah, silakan semua orang menghitung 3 ns terlebih dahulu. (Bisa diperpanjang nanti)
Jika Anda ingin mencoba perhitungan 10 ns, ubah bagian berikut:
...
nsteps = 5000000 ; Jumlah langkah MD.
nstxout = 5000 ; Koordinat dioutput setiap nstxout langkah.
nstlog = 5000 ; Frekuensi informasi ditulis ke log.
...Bandingkan relax.mdp dan continue.mdp.
3-2 Menjalankan simulasi
Kemudian, kompilasi file pengaturan. Kita akan menamai file yang dihasilkan T250.tpr.
gmx grompp -maxwarn 1 -f T250.mdp -p ice.top -c relaxed.tpr -t relaxed.cpt -o T250.tprJalankan!
gmx mdrun -deffnm T250 -nt 16Kita menentukan jumlah proses paralel dengan -nt 16. (Jika tidak ditentukan, kadang-kadang ada kasus yang menghasilkan error di tengah jalan. Tapi mungkin tidak diperlukan.) Dalam praktikum ini, kita menggunakan komputer dengan 96 core, jadi bisa menentukan hingga 96, tetapi jika jumlah proses paralel untuk semua orang melebihi 96, proses semua orang akan melambat. Harap batasi hingga 16 ketika sedang sibuk. (16 x 5 = 80 < 96)
Ini seharusnya memakan waktu 30 sampai 100 kali lebih lama dari sebelumnya. Mari kita tunggu dengan sabar sambil minum kopi.
3-3 Perpanjangan
Jika dalam analisis selanjutnya ternyata waktu simulasi masih kurang, kita bisa memperpanjang perhitungan dengan cara berikut.
Pertama, berdasarkan file .tpr yang dihasilkan oleh grompp sebelumnya, kita buat file .tpr baru dengan waktu eksekusi yang diperpanjang. Opsi -extend menentukan waktu tambahan perhitungan dalam satuan ps. Kita akan memperpanjang 2000 ps = 1 ns.
gmx convert-tpr -extend 2000 -s T250.tpr -o extended.tprKemudian jalankan! (Untuk perhitungan lanjutan, sepertinya perlu menentukan beberapa opsi.)
gmx mdrun -deffnm extended -cpi T250.cpt -s extended.tpr -noappendKali ini mungkin memakan waktu cukup lama untuk bisa makan.
4. Visualisasi hasil
Untuk mengonfirmasi apa yang sebenarnya terjadi, mari kita transfer data koordinat molekul ke komputer lokal dan tampilkan gerakan molekul menggunakan VMD.
Pertama, kita ubah data trajektori .trr menjadi format .gro.
(Anda juga bisa melakukan konversi di tengah simulasi. Dalam hal ini, buka terminal baru untuk bekerja.)
gmx trjconv -f T250.trr -s T250.tpr -pbc whole -o T250.groUnduh ini dan buka dengan VMD.
Dengan perhitungan 1 ns, mungkin sulit untuk menentukan apakah sedang meleleh atau membeku, tetapi dengan perhitungan 10 ns, biasanya bisa ditentukan.
Menurunkan suhu tidak selalu berarti pembekuan lebih cepat, karena pada suhu rendah, gerakan molekul menjadi lebih lambat, yang juga memperlambat pertumbuhan kristal.
Semakin tinggi suhu, semakin cepat meleleh, itu pasti.
5. Penilaian
Kita menyadari bahwa diperlukan waktu yang cukup lama sampai proses pertumbuhan terlihat jelas secara visual. Oleh karena itu, kita akan mengambil pendekatan yang berbeda.
Dalam simulasi dengan tekanan dan suhu konstan, kepadatan akan berbeda tergantung apakah menjadi cair atau padat. Demikian juga, energi potensial akan berbeda. Padat pasti akan memiliki energi potensial yang lebih rendah (karena entropinya lebih rendah). Dengan kata lain, dengan melihat perubahan energi potensial atau kepadatan terhadap waktu, kita dapat membedakan apakah es sedang tumbuh atau meleleh.
Mari kita plot dan periksa ini.
Perubahan waktu dari kuantitas termodinamika seperti suhu, tekanan, energi potensial, dll. selama simulasi juga dicatat dalam file .log, tetapi informasi yang lebih rinci dicatat dalam file .edr.
Perintah gmx dump digunakan untuk mengubah konten file .edr ini menjadi format yang mudah dibaca manusia.
gmx dump -e T250.edrNamun, file yang diubah dengan cara ini juga tidak terlalu nyaman untuk pemrosesan data selanjutnya. Jadi, kita akan mengubahnya lebih lanjut menjadi format tabel menggunakan skrip buatan sendiri undump.py.
gmx dump -e T250.edr | python3 undump.py > T250.txtUnduh file ini dan buka menggunakan alat yang sesuai (Excel?) untuk memplot hubungan antara waktu dan energi potensial.
5-1 Untuk Gnuplot
Kolom pertama adalah waktu, kolom ketiga adalah energi potensial, dan kolom ketujuh adalah kepadatan.
plot 'T250.txt' u 1:3 w l6. Estimasi titik leleh
Jika simulasi dilakukan cukup lama, es akan meleleh sepenuhnya pada suhu di atas titik leleh, dan akan membeku sepenuhnya pada suhu di bawah titik leleh. Dan energi potensial atau kepadatan akan mencapai nilai konstan.
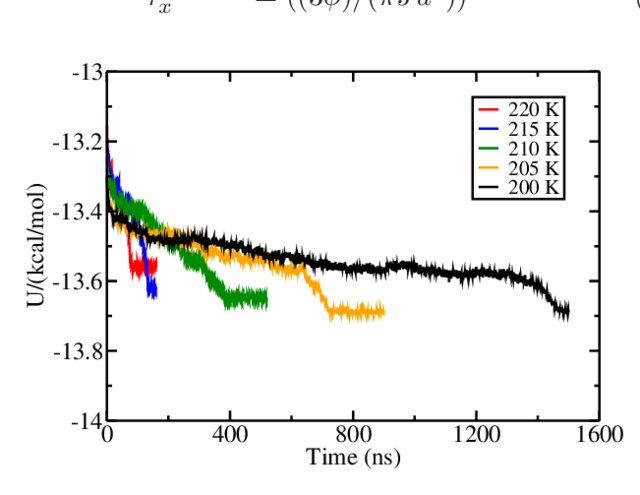
Perubahan energi potensial selama proses pertumbuhan es dari keadaan koeksistensi. Pada suhu rendah, energi yang lebih rendah tercapai tetapi membutuhkan waktu lebih lama. [^4] Titik leleh model air yang digunakan di sini diperkirakan 252 K, jadi suhu-suhu ini sesuai dengan suhu yang jauh lebih rendah dari titik leleh.
Waktu yang dibutuhkan untuk mencapai nilai konstan seharusnya menjadi lebih pendek semakin jauh dari titik leleh (namun, seperti yang disebutkan di atas, ketika suhu menjadi sangat rendah, gerakan molekul melambat sehingga membutuhkan waktu lebih lama untuk membeku.)
Misalkan waktu yang dibutuhkan untuk pembekuan/pelelehan lengkap berbanding terbalik dengan perbedaan suhu dari titik leleh. Dalam hal ini, jika kita memplot suhu pada sumbu x dan kebalikan dari waktu yang dibutuhkan untuk menyelesaikan perubahan energi potensial pada sumbu y, kita akan mendapatkan kurva untuk sisi suhu rendah dan tinggi, dan kurva-kurva ini akan mendekati 0 pada titik leleh (dengan kata lain, pada titik leleh tepat, es tidak akan membeku atau meleleh tidak peduli berapa lama kita menunggu).
Namun, ketika mendekati titik leleh, waktu yang dibutuhkan untuk pembekuan lengkap menjadi terlalu lama. Juga, jika perhitungan tidak bisa dilakukan cukup lama, kita tidak bisa mengonfirmasi pembekuan lengkap. Dalam kasus seperti itu, alih-alih menunggu energi potensial akhir diperoleh, kita bisa mengukur kecepatan penurunan/kenaikan energi potensial dan memplot kebalikannya pada sumbu y.
7. Tugas
Silakan semua orang berbagi tugas untuk mengumpulkan perubahan energi potensial terhadap waktu dari 250 K hingga 290 K (dengan interval 5 K, kecuali 270 K), dan dari hasil-hasil tersebut, perkirakan titik leleh TIP4P/Ice.
| Nama Pengguna | Suhu | Waktu Simulasi (Minimal) |
|---|---|---|
| q1 | 255 K | 10 ns |
| q2 | 285 K | 3 ns |
| q3 | 265 K | 10 ns |
| q4 | 275 K | 10 ns |
| q5 | 260 K | 10 ns |
| q6 (q1) | 280 K | 3 ns |
| q7 (q2) | 250 K | 10 ns |
| q8 (q3) | 290 K | 3 ns |
| q9 (q4) | 245 K | 10 ns |
| q10 (q5) | 240 K | 10 ns |
Menurut paper [^1] di bawah, titik leleh TIP4P/Ice adalah 270 K, tetapi dalam simulasi kali ini, kita menggunakan sistem dengan jumlah molekul yang jauh lebih sedikit untuk mempercepat perhitungan. Karena itu, ada kemungkinan titik leleh akan menyimpang dari 270 K.
Pada suhu 270 K atau suhu yang dekat dengan titik leleh yang diperkirakan, mungkin tidak akan membeku atau meleleh tidak peduli berapa lama kita menunggu.
Waktu yang dibutuhkan untuk perubahan fase selesai tidak selalu konstan. Jika ada waktu, sebaiknya melakukan beberapa kali pengukuran pada suhu yang sama dan mencari rata-ratanya.
Dalam laporan Anda, silakan tulis informasi berikut:
- Waktu yang dibutuhkan untuk pelelehan atau pembekuan pada setiap suhu (dalam format tabel) dan grafik (data mentah) yang menjadi dasar. Silakan bertukar dan berbagi data untuk suhu selain yang Anda hitung sendiri. (Jika Anda memiliki waktu lebih, Anda boleh menghitung suhu lain sendiri.)
- Grafik yang memplot kebalikan waktu yang dibutuhkan terhadap suhu. (Perkiraan kasar saja cukup)
- Penjelasan tentang bagaimana Anda memperkirakan titik leleh dari bentuk grafik 2, pertimbangan, dll.
8. Tambahan
- Jika Anda membuat es III atau es V di awal dan menentukan tekanan yang sesuai, Anda juga bisa memperkirakan titik leleh es tekanan tinggi.
- Pada tekanan tinggi, ada kemungkinan struktur kristal yang tidak diinginkan muncul dari keadaan koeksistensi. Namun, akan sulit untuk membedakannya.
- Metode yang sama bisa digunakan untuk transisi fase antara padat dan gas, atau cair dan gas.
- Untuk transisi fase antara padat dan padat, metode ini tidak bisa digunakan untuk menentukan suhu transisi karena ukuran balok sel simulasi yang disesuaikan dengan sel satuan salah satu padatan akan tidak sesuai untuk kristal lainnya. Dalam kasus seperti itu, teknik lain yang menghitung energi bebas dari perhitungan frekuensi getaran khas digunakan.
- Untuk air, transisi fase antara cairan dan fase cair lainnya juga bisa diamati![^2]
- Dalam contoh penggunaan ini, kita memilih es Ih sebagai struktur awal dan menyiapkan keadaan koeksistensi padat-cair dengan melelehkan setengahnya. Bahkan jika kita hanya mengubah tekanan menjadi tekanan tinggi, ini tidak akan menjadi simulasi keadaan koeksistensi es III atau es V dan air. Dibutuhkan waktu yang sangat lama bagi kristal untuk secara alami berubah menjadi kristal lain. Jika tekanan setinggi es V diterapkan pada es Ih, strukturnya akan runtuh dan semuanya akan menjadi cair.
- Anda juga bisa membuka beberapa terminal, menyalin folder, dan melakukan perhitungan dalam beberapa kondisi secara bersamaan.
9. Lampiran
9-1 Koneksi VPN
Akses remote ke server komputasi milik Laboratorium Kimia Teoritis Universitas Okayama (Xeon 96 core, IP 192.168.3.220). Informasi pengaturan akan diberikan secara terpisah.
Jika Anda tidak dapat terhubung dengan baik, gunakan Amazon EC2.
9-1-1 Untuk Mac
Masukkan pengaturan di System Settings->VPN->Add VPN Configuration.
https://www.cc.uec.ac.jp/ug/ja/remote/vpn/l2tp/macos114/index.html mungkin berguna sebagai referensi (silakan ganti konten yang dimasukkan sesuai kebutuhan)
9-1-2 Untuk Windows
https://www.seil.jp/saw-mpc/doc/sa/pppac/use/pppac-client/win11_l2tp.html mungkin berguna sebagai referensi (silakan ganti konten yang dimasukkan sesuai kebutuhan)
Control Panel → Network and Sharing Center → Change adapter settings → Pilih VPN → Properties → Security → Allow these protocols → Centang CHAP!!! (Fuh. Kenapa harus begitu dalam)
9-2 Penggunaan pribadi Amazon EC2
Bahkan dengan kuota gratis Amazon EC2, Anda masih bisa melakukan perhitungan yang cukup baik.
- Buat akun di AWS. Anda akan membutuhkan nomor kartu kredit.
- Di dashboard EC2, buat instans EC2. Anda dapat menggunakan tipe instans t2.micro (2 core) secara gratis.
- Pilih Ubuntu untuk OS. (Ini cocok untuk digunakan sebagai platform komputasi)
- Sebagian besar pengaturan lainnya bisa dibiarkan default, tapi tanyakan instruktur jika ada yang tidak jelas.
9-3 Instalasi Gromacs dan alat lainnya
Bahkan tanpa menggunakan cloud, Anda dapat dengan mudah menginstal Gromacs dan GenIce di sistem operasi berbasis Unix.
9-3-1 Untuk Ubuntu/Debian Linux
(Perlu dijalankan dengan hak administrator.)
apt update
apt install gromacs python3 python3-pip
pip install numpy
pip install genice29-3-2 Untuk Redhat/Amazon Linux/CentOS7
Paket gromacs tidak ditemukan di EC2 Amazon Linux/RedHat Linux. Sepertinya Anda perlu menginstal yang diperlukan secara individual dari RPM.
9-3-3 Untuk MacOS
Siapkan Homebrew terlebih dahulu.
Dengan HomeBrew, Anda dapat menginstal tanpa hak administrator.
brew install gromacs python3
pip install genice29-3-4 Untuk Windows
(Informasi dibutuhkan!)
Referensi
[^1] Conde, M. M., Rovere, M. & Gallo, P. High precision determination of the melting points of water TIP4P/2005 and water TIP4P/Ice models by the direct coexistence technique. J. Chem. Phys. 147, 244506 (2017).
[^2] Yagasaki, T., Matsumoto, M. & Tanaka, H. Spontaneous liquid-liquid phase separation of water. Phys. Rev. E Stat. Nonlin. Soft Matter Phys. 89, 020301 (2014).
[^3] Yeyue Xiong, Parviz Seifpanahi Shabane, and Alexey V. Onufriev*, Melting Points of OPC and OPC3 Water Models, ACS Omega 39, 25087–25094 (2020).
[^4] Espinosa, J. R., Sanz, E., Valeriani, C. & Vega, C. Homogeneous ice nucleation evaluated for several water models. J. Chem. Phys. 141, 18C529 (2014).